Alphafold2蛋白质结构预测AI工作站配置推荐24v1
价格变更日期:2024/01/05(2024年第1季度),
变更原因:GPU部分价格变化,CPU部分升级换代
目录
AlphaFold2计算特点
蛋白质三维结构预测AI工作站配置推荐
蛋白质三维结构预测AI集群配置推荐
蛋白质三维结构预测是一项计算量非常巨大的任务,科学家多年的探索研究,形成了X射线晶体学法、核磁共振法、冷冻电镜等
2021年底,谷歌的DeepMind团队的采用人工智能方法的AlphaFold2算法在生物界引起了极大的轰动,它能准确地预测蛋白质的结构,AlphaFold2是当今预测蛋白质3D结构的最强工具。它将被大量用于推动世界蛋白质研究向前发展.
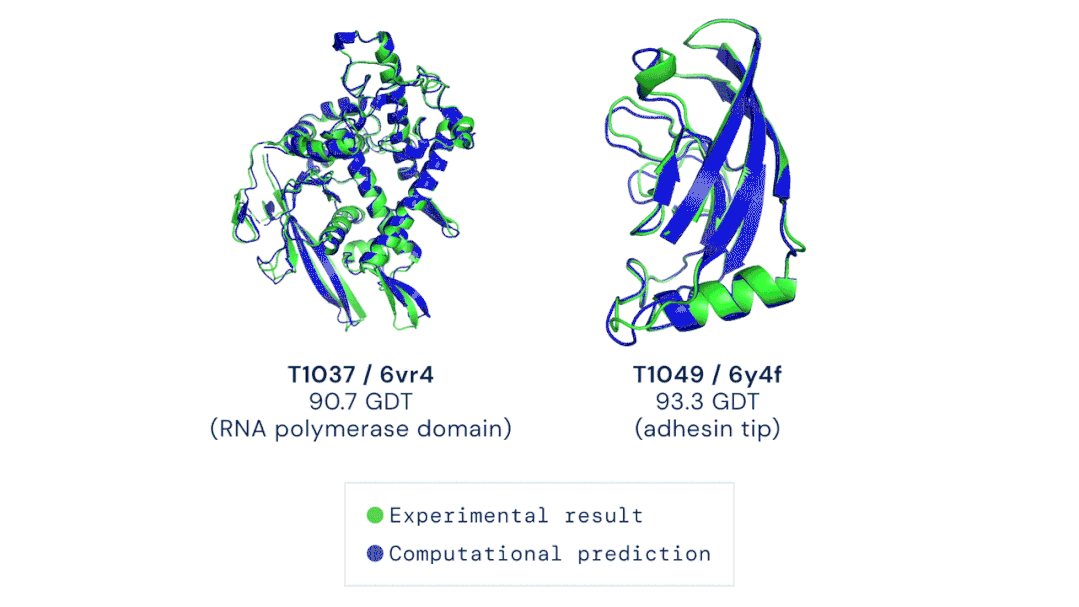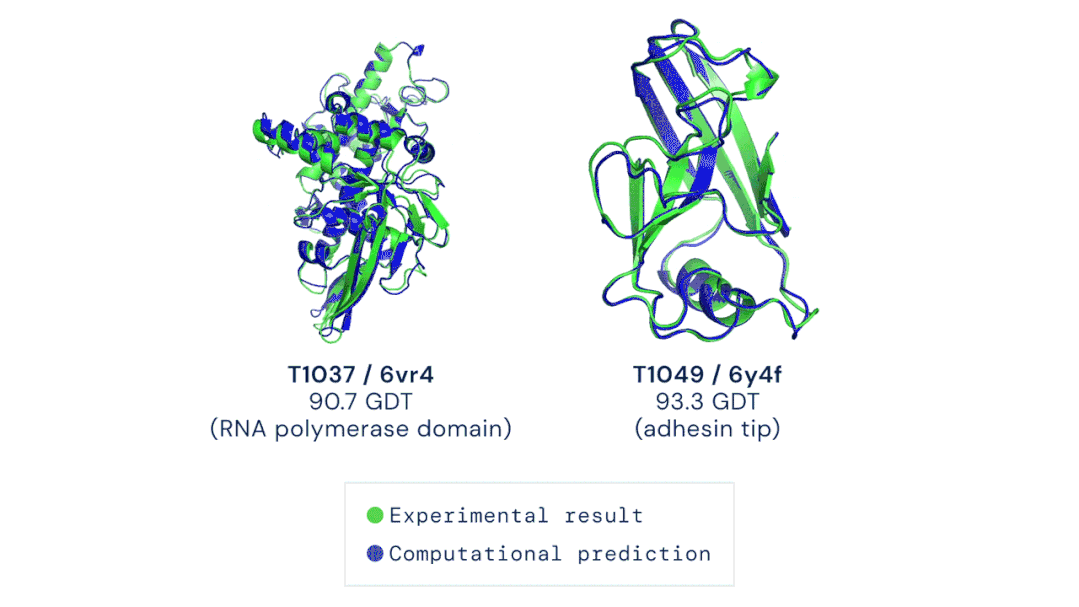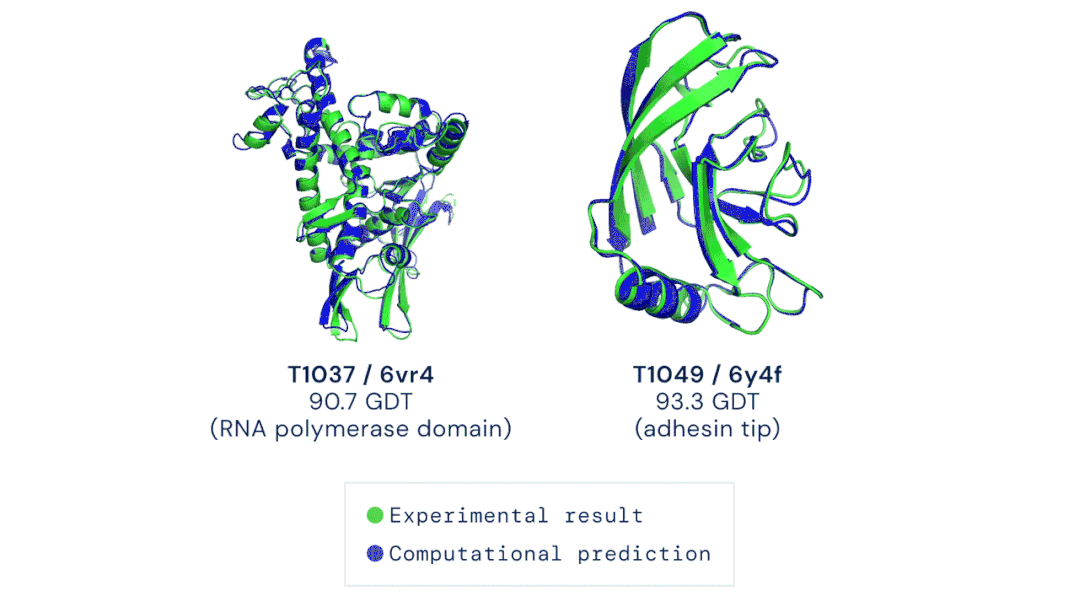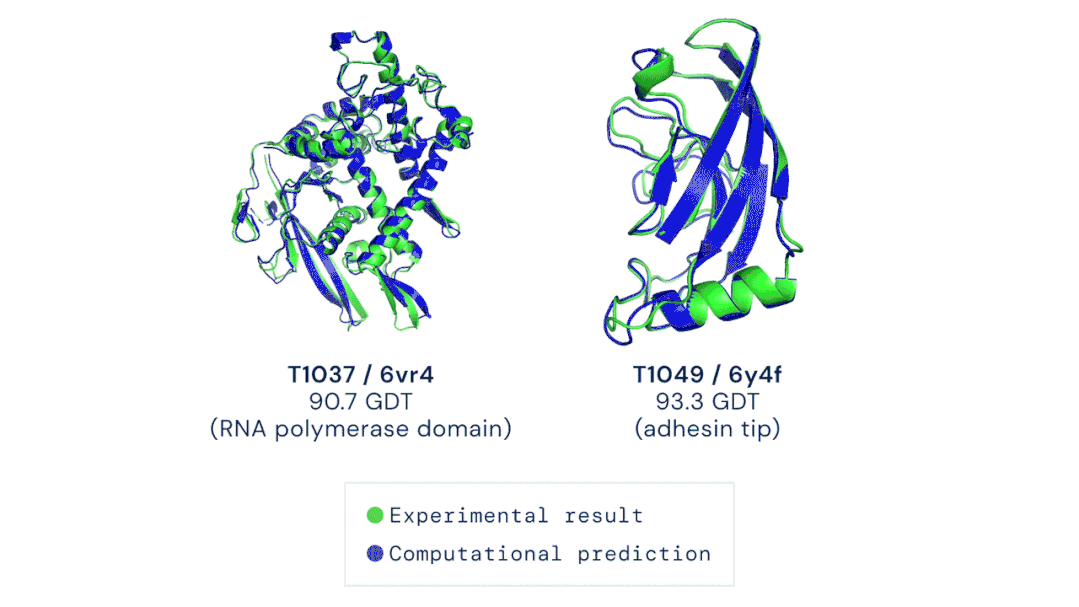
AlphaFold2在国际蛋白质结构预测竞赛(CASP14)上精确地基于氨基酸序列预测蛋白质的3D结构。其准确性可以与使用冷冻电子显微镜(CryoEM)、核磁共振或 X 射线晶体学等实验技术解析的3D结构相媲美,
目前情况(大致统计):
(1)Deepmind开源了AlphaFold2的源代码(推理部分)
(2)华盛顿大学开源了RoseTTRFold的源代码(推理部分)
(3)深势科技复现了AlphaFold的训练部分,并开源代码(训练和推理)
(4)上海天壤智能科技有限公司复现了TRfold训练部分和推理部分
(5)上海交大对AlphaFold2的推理代码进行了优化(推理并行版)
(一)AlphaFold2蛋白质结构预测计算特点
如何配置好硬件,最快速度完成训练、推理计算,首先分析其计算过程以及算法特点
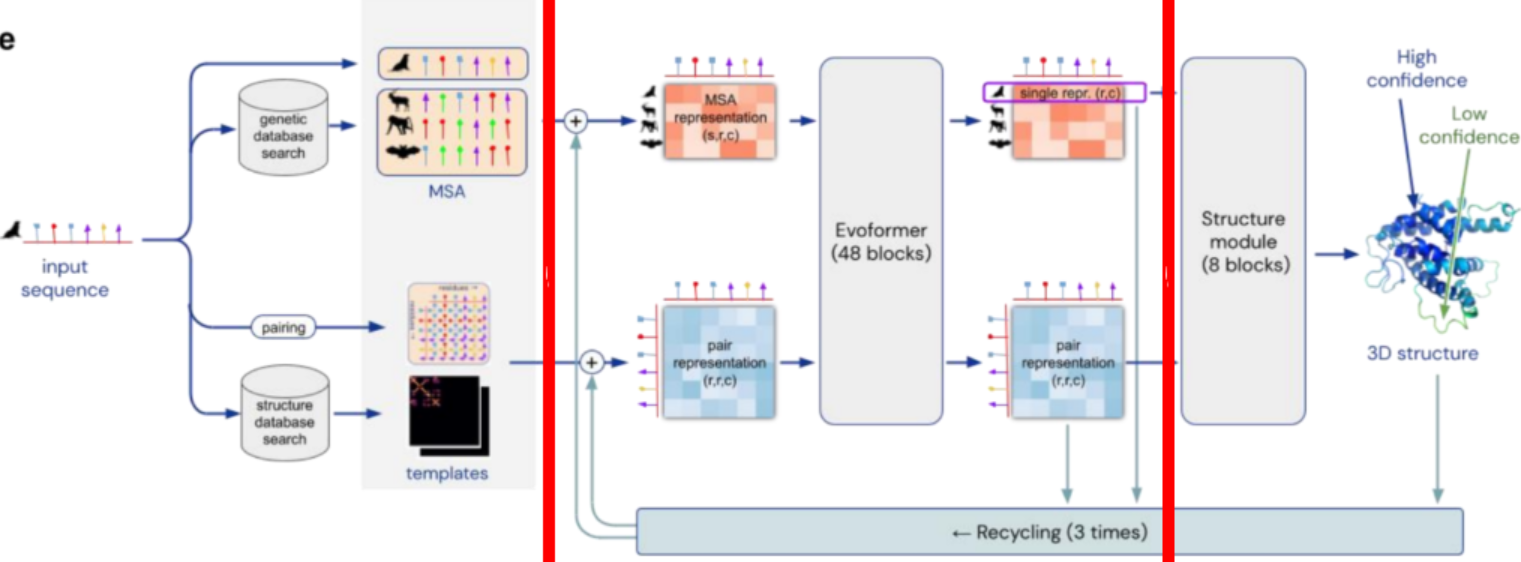
图1 Alphafold2计算示意图
环节1 数据处理-序列特征生成计算特点
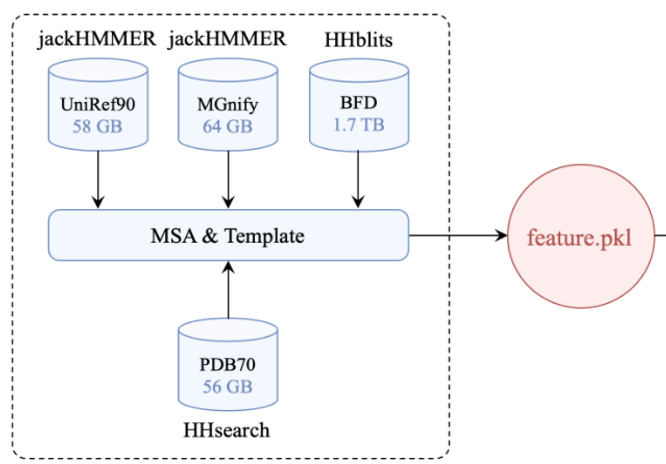
(图片2来源:上海交大alphafold2并行优化版)
计算过程
总输入单个蛋白质序列FASTA格式(推理);
通过搜索工具(jackHMMER/HHblits)分别对多个遗传数据库--执行隐马尔可夫模型的搜索生成MSA(序列-残基);见图1
搜索的结构和序列产生的Pairing信息(残基-残基);
通过HHsearch搜索的Template
计算与硬件配置分析
数据库搜索过程涉及数据库密集I/O读写,数据放到高速SSD硬盘上,数据量累积超过2TB,非常耗时,加速手段提升CPU计算速度。
硬件配置
CPU计算为主,内存要够大,或配备NVME SSD固态卡,容量4TB以上
环节2 神经网络预测计算特点
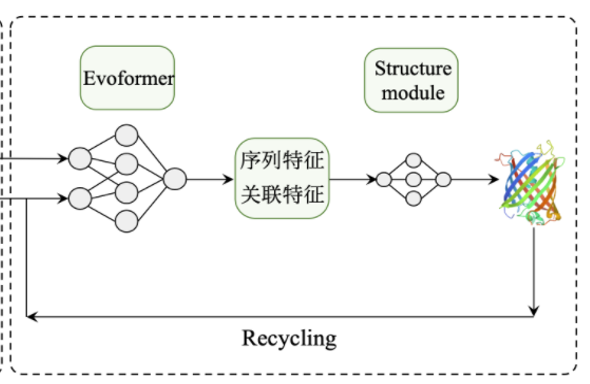
(图片来源:上海交通大学 https://parafold.sjtu.edu.cn/docs/quick-start/)
计算过程
利用多序列比对(MSA),把蛋白质的结构和生物信息整合到深度学习算法中,主要包括:神经网络EvoFormer和结构模块(Structure module).
在EvoFormer中,主要是将图网络(Graph networks)和多序列比对(MSA)结合完成结构预测,Alphafold2使用Transformer结构,不管是MSA还是残基-残基对的信息更新都使用了Attention机制,结构模块的更新使用了三角法则,简化了计算的复杂度,准确率也提高了不少.
结构模块(Structure Module)主要工作是将EvoFormer得到的信息转换为蛋白质3D结构.
整个模型的Evoformer和Structure module部分都使用了Recycling,即将输出重新加入到输入在重复refinement,进行信息的精炼.
计算特点
上述计算过程用GPU更合理,对GPU要求是高显存带宽、大容量显存、大蛋白质计算通过将多GPU卡设置统一内存架构,大的显存可支持更大的计算数据存放
计算架构分析汇总
|
No |
主要环节 |
任务分类 |
计算时间占总求解时间比例 |
计算特点 |
配置方案 |
|
1 |
序列特征生成 |
(a)多序列比对 (b)模板搜索 |
计算时间占总计算时间的大约60% |
CPU计算 有限多核
|
高频CPU加速 4TB容量的NVME SSD盘 |
|
2 |
神经网络预测 |
(c)模型预测 (d)AMBER优化 |
计算时间占总计算时间的大约40% |
单GPU计算(推理) 多GPU计算(训练)
|
显存容量大,支持更长序列蛋白计算(最大长度1500,大于300个残基) 多卡统一内存模式 |
#p#page_title#e#
(二)蛋白质结构预算AI工作站配置推荐2024v1
西安坤隆计算机科技有限公司专注于工作站专业应用,对每个应用的计算过程研究分析,给出精准高效、高可靠计算架构和专业系统优化,以及稳定的技术支持,保证与应用软件90%以上的匹配吻合,和长期稳定运行.

硬件配置具有以下特点:
1)配备CPU规格均以高频为主,兼顾足够CPU核数,这样保证数据预处理(最慢的环节),计算时间大幅缩短,GPU卡采用单精度指标高的、显存容量大的,保证神经预测计算加速,满足AlphaFold2的理想的配置方案
2)整机的cpu、gpu、硬盘配置,满足AlphaFold2推理(包括上海交大并行版)计算要求,均衡无死角,性能最大化;
3)为深势科技的Uni-Fold训练、推理模块提供理想配置架构;
4)每个配置机器做到即开即用,并提供硬件+优化+稳定高速运行技术支持服务
5)支持大规模的计算扩展应用需求
2.1 蛋白质结构预测工作站配置参考24v1
NO
品牌与型号
配置规格
价格
1
GA330i 15764-MCT
intel 14代高频处理器(共计24核,8核@5.7GHz,16核@4.2GHz /64GB DDR5/ RTX4090 24GB /960GB SSD/3.84TB PCIe.SSD/6TB SATA企业级/微塔式(1500w)/24寸-2K显示器
56000
AF2推理型
2
GA330i 157128-MCE
intel 14代高频处理器(共计24核,8核@5.7GHz,16核@4.2GHz /128GB DDR4/A6000 48GB/960GB SSD/3.84TB PCIe.SSD/8TB SATA企业级/微塔式(1500w)/24寸-2K显示器
81000
AF2推理顶配型
3
EX650i 228192-MCT
2*Xeon 银4316处理器(共计40核,2.8GHz~3.4GHz) /192GB DDR4 /A6000 48GB/960GB SSD /3.84TB PCIe.SSD/8TB SATA企业级/微塔式(1500w)/24寸-2K显示器
86500
并行优化加速型
4
GX650M 228192-MC2T
2*Xeon 银4316处理器(共计40核,2.8GHz~3.4GHz) /192GB DDR4 /2*RTX4090 24GB/480GB SSD /3.84TB PCIe.SSD/16TB SATA企业级/微塔式(1500w)/24寸-2K显示器
112000
多任务高速型
5
GX650M 228256-MC2E
2*Xeon 银4316处理器(共计40核,2.8GHz~3.4GHz)/256GB DDR4/2*A6000 48GB/1TB SSD /3.84TB PCIe.SSD/16TB SATA企业级/微塔式(1500w)/24寸-2K显示器
149990
多任务大规模计算型
6
GX650M 228512-MC4T
2*Xeon 银4316处理器(共计40核,2.8GHz~3.4GHz)/512GB DDR4 /4*RTX4090 24GB /1TB SSD /7.68TB PCIe.SSD/16TB SATA企业级/双塔式(2000w)/24寸-2K显示器
197000
多任务高速加强型
7
GX650M 231384-MC4E
2*Xeon 金6326处理器(共计32核,3.1GHz )/384GB DDR4/4*A6000 48GB /1TB SSD /7.68TB PCIe.SSD /2*16TB SATA企业级/双塔式(2000w)/24寸-2K显示器
285000
全能加速型
8
GX650M 231768-MC6E
2颗Xeon 金6342处理器(共计48核,3.1GHz)/768GB DDR4/6*A6000 48GB /2TB SSD/7.68TB PCIe.SSD /2*18TB SATA企业级/双塔式(2000w)/24寸-2K显示器
399990
推理/训练多任务高速型
9
GX650M 2351T-MC8E
2颗Xeon 铂金8358处理器(共计64核,3.5GHz)/1TB DDR4/8*A6000 48GB /2TB SSD/15.36TB PCIe.SSD /2*18TB SATA企业级/双塔式(2000w)/24寸-2K显示器
580000
推理/训练极致型
2.2 AlphaFold训练集群配置参考23v1
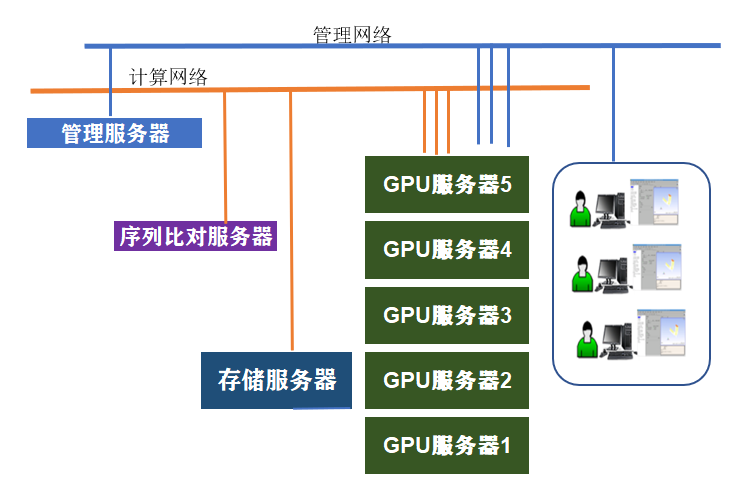
集群技术特点
(1)本集群是由高频服务器、GPU计算服务器、管理服务器、并行存储服务器组成。
(2)集群硬件配置每个环节都基于人工智能预测蛋白质三维结构算法最快优化设计。
(3)【高频服务器】采用有限多核高频CPU、高速NVME SSD用于密集海量序列比计算,计算性能和io读写性能完美匹配高效,支持多个单核计算或2个以上并行版的比对软件同步计算。
(4)【GPU计算服务器】配备最新Xeon3代处理器,32核+8块RTX A6000 48GB,cpu频率和pcie 4.0 x16是蛋白质折叠人工智能并行计算最理想架构。
(5)作业调度软件针对蛋白质预测元计算两个主要环节:序列比对计算(CPU有限多核计算环节)、人工智能预测(GPU多卡并行计算环节),
序列比对计算环节--调用【高频服务器】处理,
蛋白质结构预测计算环节--调用多台【GPU服务器】并行计算。
(6) 集群性能从硬件架构、作业调度系统均比常规集群性能达到极致,全新改写的作业调度系统,比市面上的作业调度系统效率更高。
方案1 蛋白质智能预测集群方案(40块GPU卡)
|
NO |
型号 |
技术规格 |
数量 |
单价 |
小计 |
|
1 |
序列比对服务器 |
intel 14代超频处理器 (共计24核,8核@5.7GHz,16核@4.2GHz)/192GB DDR5/960GB/4U机架式(650w)/25GbE网口 |
1 |
28000 |
28000 |
|
2 |
GPU超算服务器 |
2颗Xeon 银4314处理器 (32核@3.5GHz)/512GB/8块A6000 /960GB/4U机架式(2000w 2+2冗余/100GbE网口 |
5 |
450000 |
2250000 |
|
3 |
并行存储服务器 |
2颗Xeon 银4314处理器 (24核@2.1GHz)/128GB /960GB /45TB高速缓存盘(全闪存阵列,读20GB/s、写11GB/s)/252TB并行存储(双通道)/4U机架式(1200w 1+1冗余/100GbE网口 |
1 |
165000 |
165000 |
|
4 |
管理服务器 |
2颗Xeon 银4210 /128GB /RTX A2000/1.92TB/2U机架式/千兆 |
1 |
32000 |
32000 |
|
5 |
计算交换机 |
MSN2010 4*100G/18*25G,2U机架式 |
1 |
45000 |
45000 |
|
6 |
管理交换机 |
24口千兆,1U机架式 |
1 |
2800 |
2800 |
|
7 |
KVM切换器 |
16口 VGA,1U机架式 |
1 |
1800 |
1800 |
|
8 |
机柜 |
42U |
1 |
5500 |
5500 |
|
9 |
集群系统 |
UltraLAB PSS作业调度管理软件(5节点) |
1 |
50000 |
50000 |
|
|
|
|
|
|
|
|
|
合计 |
|
¥2,580,100元 |
||
方案2 蛋白质智能预测集群方案(80块GPU卡)
|
NO |
型号 |
技术规格 |
数量 |
单价 |
小计 |
|
1 |
序列比对服务器 |
Xeon W7-2465X超频处理器 (共计28核4.8GHz)/256GB DDR5/960GB/4U机架式(850w) /25GbE网口 |
1 |
63000 |
63000 |
|
2 |
GPU超算服务器 |
2颗Xeon 银4314处理器 (32核@3.5GHz)/512GB/8块A6000 /960GB/4U机架式(2000w 2+2冗余/100GbE网口 |
10 |
450000 |
4500000 |
|
3 |
并行存储服务器 |
2颗Xeon 银4314处理器 (24核@2.1GHz)/128GB /960GB /45TB高速缓存盘(全闪存阵列,读20GB/s、写11GB/s)/252TB并行存储(双通道)/4U机架式(1200w 1+1冗余/100GbE网口 |
1 |
165000 |
165000 |
|
4 |
管理服务器 |
2颗Xeon 银4210 /128GB /RTX A2000/1.92TB/2U机架式/千兆 |
1 |
32000 |
32000 |
|
5 |
计算交换机 |
MSN2010 4*100G/18*25G,2U机架式 |
1 |
45000 |
45000 |
|
6 |
管理交换机 |
24口千兆,1U机架式 |
1 |
2800 |
2800 |
|
7 |
KVM切换器 |
16口 VGA,1U机架式 |
1 |
1800 |
1800 |
|
8 |
机柜 |
42U |
2 |
5500 |
5500 |
|
9 |
集群系统 |
UltraLAB PSS作业调度管理软件(10节点) |
1 |
100000 |
100000 |
|
|
|
|
|
|
|
|
|
合计 |
|
¥4,920,600元 |
||
参考资料:
AlphaFold2 https://github.com/deepmind/alphafold/
上海交大 AlphaFold再HPC平台的部署和优化
https://parafold.sjtu.edu.cn/docs/quick-start/
PombertLab/3DFI
https://github.com/PombertLab/3DFI
上述所有配置,代表最新硬件架构,同时保证是最完美,最快,如有不符,可直接退货
欲咨询机器处理速度如何、技术咨询、索取详细技术方案,提供远程测试,请联系
UltraLAB图形工作站供货商:
西安坤隆计算机科技有限公司
国内知名高端定制图形工作站厂家
业务电话:400-705-6800
咨询微信号:










